A02班
クロススケール細胞内分子構造動態解析が解明する小胞体恒常性維持機構


渡部 聡(研究分担者)
東北大学 多元物質科学研究所 助教
計画研究の概要
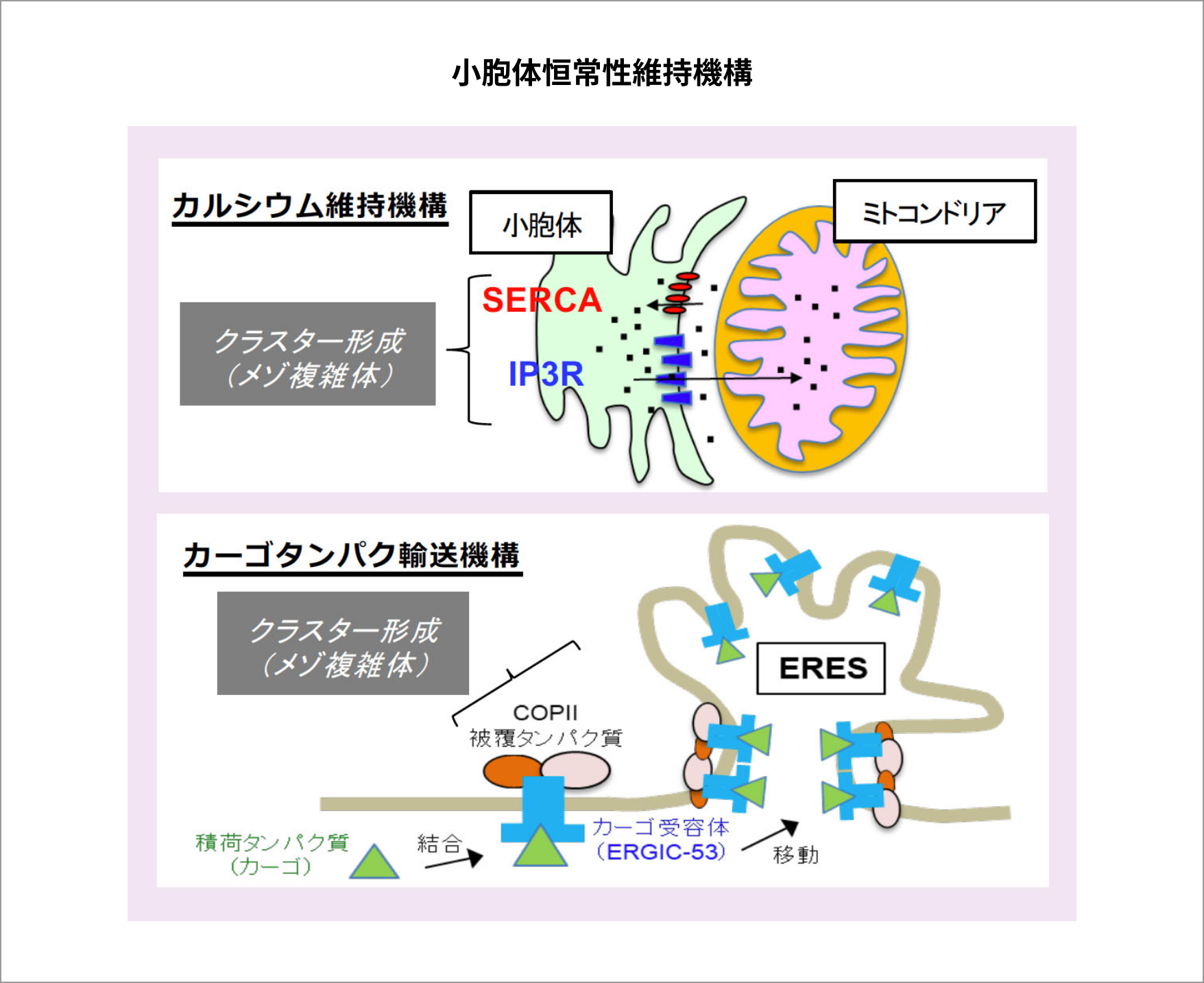
細胞内には、新規合成タンパク質の立体構造や局在、さらにはレドックス、pH、金属イオンなどの化学パラメータを厳密に制御するためのシステムが存在します。本研究課題では、カーゴ受容体による積荷タンパク質輸送の分泌初期過程、および小胞体におけるカルシウム恒常性維持に関わるイオンポンプ・チャネルに焦点をあて、それら因子の “細胞内環境” での作用機序解明を目指します。具体的には、吉川Gが有するクライオ電子線トモグラフィー技術、福間Gが有するIn-cell AFM技術、水上Gが有するオルガネラ内の化学パラメータを可視化定量および一分子超解像イメージング技術、さらには杉田Gが有する細胞内環境を考慮に入れた大規模分子動力学計算技術等を統合し、上記課題を深く究明します。以上の研究をとおし、現在もなお国内外の多くの研究者が活発に進める小胞体恒常性維持機構の研究分野に大きなパラダイムシフトを引き起こすことを目指します。
主な研究業績
- Zhang, Y., Watanabe, S., Tsutsumi, A., Kadokura, H., Kikkawa, M. and Inaba, K. “Cryo-EM analysis provides new mechanistic insight into ATP binding to Ca2+-ATPase SERCA2b” EMBO Journal 40, e108482 (2021)
- Zhang. Y., Inoue, M., Tsutsumi, A., Watanabe, S., Nishizawa, T., Nagata, K., Kikkawa, M. and Inaba, K. “Cryo-EM structures of SERCA2b reveal the mechanism of regulation by the luminal extension tail” Science Advances 6, eabb0147 (2020)
- Kadokura, H., Dazai, Y., Fukuda, Y., Hirai, N., Nakamura, O., and Inaba, K. “Observing the nonvectorial yet cotranslational folding of a multidomain protein, LDL receptor, in the ER of mammalian cells” Proc Natl Acad Sci U S A. 117, 16401-16408 (2020)
- Inoue, M., Sakuta, N., Watanabe, S., Zhang, Y., Yoshikaie, K., Tanaka, Y., Ushioda, R., Kato, Y., Takagi, J., Tsukazaki, T., Nagata, K. and Inaba, K. “Structural basis of sarco/endoplasmic reticulum Ca2+-ATPase 2b regulation via transmembrane helix interplay” Cell Reports, 27, 1221-1230 (2019)
- Watanabe S.#, Amagai Y.#, Sannino S.#, Tempio T., Anelli T., Harayama M., Masui S., Sorrentino I., Yamada M., Sitia R, and Inaba K. (#equal contribution) “Zinc regulates ERp44-dependent protein quality control in the early secretory pathway.” Nature Communications, 10, 603 (2019)
- Okumura, M.#, Noi, K.#, Kanemura, M., Kinoshita, M., Saio, T., Inoue, Y., Hikima, T., Akiyama, S., Ogura, T. and Inaba, K. (#equal contribution) “Dynamic assembly of protein disulfide isomerase in catalysis of oxidative folding” Nature Chemical Biology 15, 499-509 (2019)